vcf2phylip.pyの使い方-VCFをphy/fasta/nexusに変換する
vcf2phylip.py
使用難易度★☆☆☆☆
今回は何をする?
VCFファイルを系統樹作成に使用するnexusやfasta、phy形式に変換できるプログラムです
簡単・短時間で実行できるので、SNPデータを使った系統解析を行う際に大活躍します。
Twitterで記事の更新をお知らせしているので、興味を持たれた方は是非フォローをお願いします。
フォローする @harrykun_blog
vcf2phylip.pyの詳細はGithubのリンクからどうぞ
続きを読むdos2unixの使い方-知らないと損するツール集 Part1 [dos2unix]
ゲノム解析にちょこっと役立つ、でも知らないと損するツールを箇条書き程度の情報量で紹介します。
dos2unix
Twitterで記事の更新をお知らせしているので、興味を持たれた方は是非フォローをお願いします。
フォローする @harrykun_blog
CRLFテキストをUnix形式に変換するツール
ワードやエクセルで作成したテキストファイルをプログラムで使用すると、改行コードがWindow用のCRLF形式で保存されているためにエラーが出ることが頻繁にある。
dos2unixを使って、ファイル形式をASCII形式に変換する。
使用方法の例
インストールする。
conda install -c conda-forge dos2unix
入力ファイルにエラーが出た際に、fileコマンドで元のファイル形式を確認する。
file XXXX.txt ASCII text, with CRLF line terminators
のようにCRLFとなっているので、これを変換する。
dos2unix XXX.txt
これだけで、ASCII textに変換される。
今回はこれで終わりです。
よければ他の記事のも見ていってください。
バイオインフォマティクス関連の書籍紹介は↓こちら
eupatho-bioinfomatics.hatenablog.com
VCFファイルとはなにかを説明します-後編
VCF (Variant call format)ファイルの見方
今回は何をする?
本記事は、VCFファイルを解説する記事の後編です。
前半をご覧になっていない方はこちらからどうぞ。
後編では各バリアントサイトのレコードに付随する項目やスコアについて説明します。
GATK公式HPのVCFについてのリンク
今回はこちらで用意したVCFファイルをもとに説明していきます。すべてを貼り付けると長大になってしまうため、一部を抜粋します。 全体を見たい方はこちらのリンクからダウンロードしてください。(400MBほど) メインコンテンツのGATK解説シリーズで、このVCFファイルを得る手順を詳しく解説していますので、興味を持たれた方はそちらものぞいてみてください。
eupatho-bioinfomatics.hatenablog.com
Twitterで記事の更新をお知らせしているので、興味を持たれた方は是非フォローをお願いします。 フォローする @harrykun_blog
eupatho-bioinfomatics.hatenablog.com
1. バリアントサイトの各項目について
各バリアントサイトのレコードは、次の10個の要素で構成されています。
#CHROM POS ID REF ALT QUAL FILTER INFO FORMAT sample-nameが続く・・・ .
1行だけ抜粋したものを下に記載します。
#CHROM POS ID REF ALT QUAL FILTER INFO FORMAT ERR015377 Pf3D7_01_v3 110 . A C 124.72 . AC=1;AF=0.071;AN=14;DP=99;FS=0.000;MLEAC=2;MLEAF=0.143;MQ=36.03;QD=31.18;SOR=3.258 GT:AD:DP:GQ:PL 0:9,0:9:99:0,135
それでは順番に見ていきましょう。
CHROMとPOS
#CHROM POS Pf3D7_01_v3 110 Pf3D7_01_v3 260 Pf3D7_01_v3 264
バリアントがあるコンティグとゲノムの座標情報です。
欠失の場合は、実際にはその前の塩基が指定されることに注意。
ID
#CHROM ID Pf3D7_01_v3 . Pf3D7_01_v3 . Pf3D7_01_v3 .
バリアントの識別子。参照したデータベースに、このサイトのレコードが存在すると記載されます。
情報がない場合は、「・」で表記されます。
典型的な識別子はdbSNPのIDで、ヒトのデータではrs28548431のようになります。
REFとALT
#CHROM REF ALT Pf3D7_01_v3 A C Pf3D7_01_v3 C CTCTTACTTACTTACT
参照遺伝子と対立遺伝子が記載されています。そのバリアントがSNPなのかインデルなのかを教えてくれます。上記の例の場合、一つ目がSNPで、二つ目がインデルです。
QUAL
#CHROM QUAL Pf3D7_01_v3 124.72 Pf3D7_01_v3 251.07 Pf3D7_01_v3 248.82
シーケンスデータから、この部位にREF/ALT多型が存在する可能性をPhred-scaledで表したものです。Phred-scaledについてはこちらを参照してください。ちなみに10=10分の1、20は100分の1のエラー確率を表します。ただし、この値は大量のサンプルを用いてバリアントコールを行うと、非常に大きくなる可能性があるため、QUALはバリアントコールの品質を評価するためにはあまり有用な数値ではありません。
VCFファイルとはなにかを説明します-前編
VCF (Variant call format)ファイルの見方
今回は何をする?
- これまでに全くVCFファイルに触れたことのない方に向けて、ファイルを構成する要素を詳しく解説します。
- VCFは変異解析に不可欠な要素であり、必ず理解しなければなりません。 初めて見た時の感想は、「うわ、ちんぷんかんぶん・・・」という印象を持つと思いますが、慣れてくると意外と単純な要素で構成されていることに気が付きます。VCFファイルの形式を理解しないと、GATKやvcf/bcftoolsを使ったフィルタリングで何をしているか理解できなくなってしまうので、この機会に一緒に勉強していきましょう。
- 二つの記事を読み終わる頃には、きっとVCFファイルに対する苦手意識は無くなっていると思います!前編では、VCFを構成する要素について順番に解説します。後編では各バリアントサイトのレコードに付随する項目やスコアについて説明します。
Twitterで記事の更新をお知らせしているので、興味を持たれた方は是非フォローをお願いします。 フォローする @harrykun_blog
今回はこちらで用意したVCFファイルをもとに説明していきます。すべてを貼り付けると長大になってしまうため、一部を抜粋します。 全体を見たい方はこちらのリンクからダウンロードしてください。(400MBほど)
GATK公式HPのVCFについてのリンク
1. VCFフォーマットとは
VCF(Variant Call Format)を一言で説明すると、SNP、インデル、構造的変異を記述するために標準化されたテキストファイル形式です。
フォーマットの詳細な仕様は、こちらのPDFで見ることができます。
VCFはGATKがバリアントコールに使用しているフォーマットで、主にバリアントの種類とシークエンス、および個々のバリアントに関する複数サンプルのジェノタイプについての情報が記述されます。とはいえ、GATK HaplotypeCallerのようなツールが生成するVCFファイルは少し複雑です。ここでは、GATKが出力するVCFファイルを理解するために知っておくべき点について説明します。
なお、VCFファイルはプレーンテキストファイルなので、Excelなどのテキストエディタで開いて編集することができますが、VCFファイルは大きな容量になりがちであるためファイルを読み込むのに時間がかかる場合があります。そのため、GATKのSelectVariantsのようなツールを使ってデータを編集するアプローチをとる必要がでてくるわけです。
また、Microsoft Wordなどのワープロでvcfを編集するとフォーマットが崩れてしまうので絶対にしないでください。
2. VCFファイルの構成
VCFファイルは、ヘッダーとバリアントコールレコードの2つの要素で構成されています。
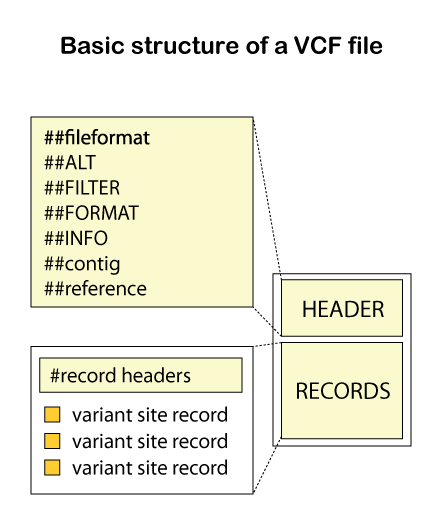
実際のデータを順番に見ていきましょう。
続きを読む