GATKによる変異検出のためのロードマップ [GATK解説シリーズのまとめ記事]
GATKの使い方 BAMファイルからVCF出力までのロードマップ
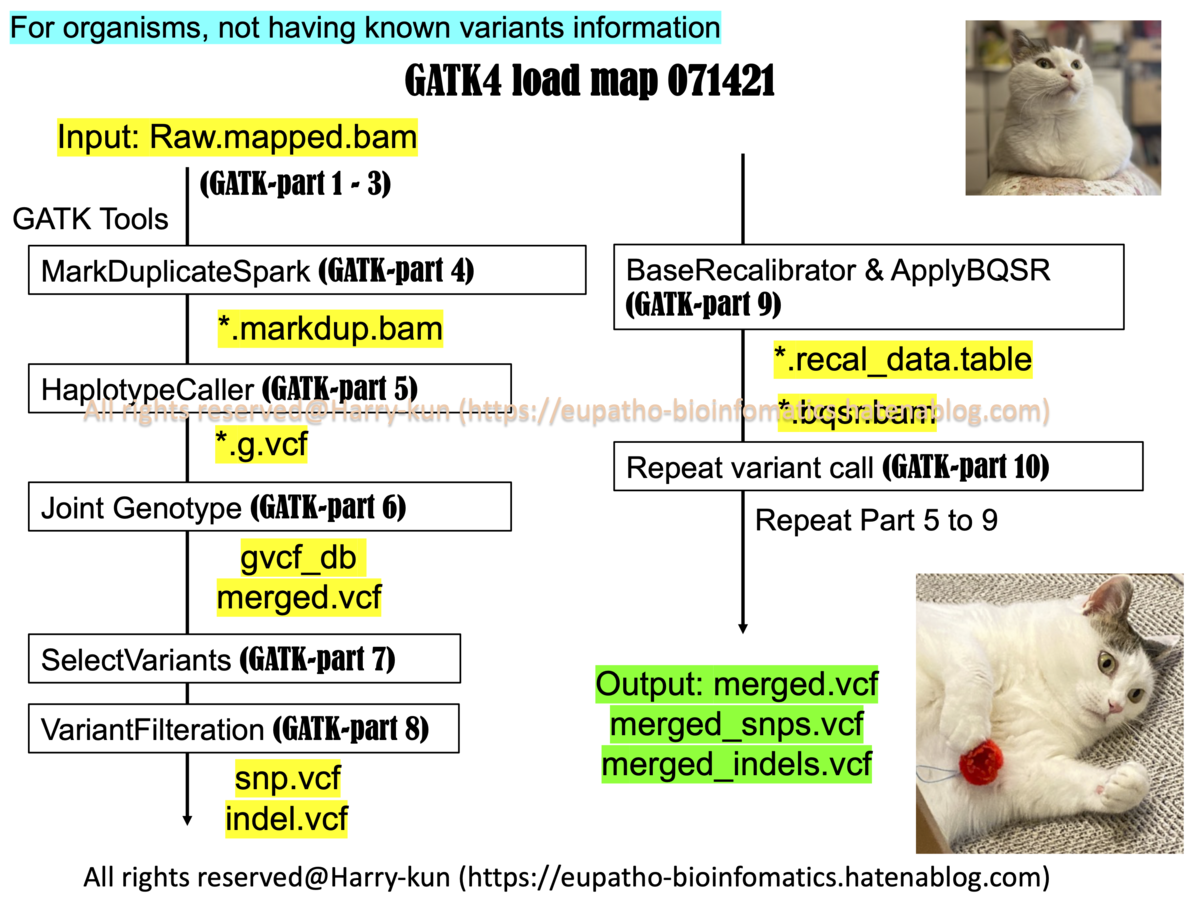
GATK4.2の使い方について、ロードマップを作成しました。
各partに対応した作業内容について、1つずつ記事にしています。
ちなみに、ブログ主の研究対象がハプロイドの病原体なので、とりあえず1倍体の生物を対象にしています。
いつになるかわかりませんが、ヒト(2倍体ゲノム)を使った内容も勉強して紹介したいと考えています。
新しい関連記事を投稿するごとに順次内容を更新しますので、本ブログでGATKに関する記事を検索したい方はこのページをブックマークされておくと便利に使っていただけると思います。
Twitterで記事の更新をお知らせしているので、興味を持たれた方は是非フォローをお願いします。
フォローする @harrykun_blog
GATK解説シリーズ パート1 GATKの導入
GATKはBroad研究所が提供する、変異の検出に特化したゲノム解析ツールキットです。非常に多機能でゲノム解析分野で業界標準として広く使われていますが、操作難易度が高く、初心者は必ず躓くポイントになっています。GATKはもともとヒトゲノム解析用に開発されたものですが、現在ではあらゆる生物のゲノムデータを扱えるように進化しています。その範囲は、体細胞・生殖細胞のショートバリアント/インデル検出、コピー数(CNV)や構造変異(SV)への解析に及びます。また、GATKには、WGSの処理や品質管理などの関連作業を行うための多くのユーティリティが含まれており、頻繁に使われるPicardも搭載されています。また、GATKの読み方は(ジー・エー・ティー・ケイ)です。公式HPはこちら
eupatho-bioinfomatics.hatenablog.com
GATK解説シリーズ パート2 使用するWGSデータの収集
今後の解説で例として使用するマラリア原虫のWGSデータを収集します。ここでは。fasterq-dumpを使ったダウンロードの方法を紹介します。
この後は、GWASで使用する変異データの取得までを順番に紹介してきます。
eupatho-bioinfomatics.hatenablog.com
GATK解説シリーズ パート3 今後のGATK解析で使用するWGSデータのマッピング
前回の記事で収集したマラリア原虫のWGSデータを使ってマッピングを行い、BAM形式のファイルを生成します。
本記事では、トリミングからマッピングまでの工程をコンパクトに記載しているので、実際にご自身のデータを使って再現される場合には、この記事のスクリプトを置き替える形で使ってもらえると便利だと思います。
eupatho-bioinfomatics.hatenablog.com
GATK解説シリーズ パート4 GATK MarkDuplicatesの使い方
- 前回の記事で得たBAM形式ファイルを使って、GATK MarkDuplicates/MarkDuplicateSparkにより重複したリードにタグを付けます。
eupatho-bioinfomatics.hatenablog.com
GATK解説シリーズ パート5 GATK HaplotypeCallerの使い方
GATK HaplotypeCallerを使って、前回の記事で得たBAM形式ファイルから、変異情報の記載されたVCFファイルを出力します。
前編では、GATK HaplotypeCallerについて解説しました。
後編では、GATK HaplotypeCallerを実践します。
eupatho-bioinfomatics.hatenablog.com
eupatho-bioinfomatics.hatenablog.com
番外編 VCFとはなにかを説明します
- これまでに全くVCFファイルに触れたことのない方に向けて、ファイルを構成する要素を詳しく解説します。
- VCFは変異解析に不可欠な要素であり、決して避けては通れません。 初めて見た時の感想は、「うわ、ちんぷんかんぶん・・・」という印象を持つと思いますが、慣れてくると意外と単純な要素で構成されていることに気が付きます。VCFファイルの形式を理解しないと、GATKやvcf/bcftoolsを使ったフィルタリングで何をしているか理解できなくなってしまうので、この機会に一緒に勉強していきましょう。
- 二つの記事を読み終わる頃には、きっとVCFファイルに対する苦手意識は無くなっていると思います!
前編では、VCFを構成する要素について順番に解説しました。
後編では各バリアントサイトのレコードに付随する項目やスコアについて説明しました。
eupatho-bioinfomatics.hatenablog.com
eupatho-bioinfomatics.hatenablog.com
GATK解説シリーズ パート6GATK JointGenotypingの使い方
- GATK GenomicsDBimport および GATK GenotypeGVCFs を使ったJoint Genotypingを実施して、複数のvcfファイルをまとめたmerged.vcfファイルを出力します。
eupatho-bioinfomatics.hatenablog.com
GATK解説シリーズ パート7 GATK SelectVariantsの使い方
- GATK SelectVariantsを使って、merged.vcfファイルから、自身の解析条件に適したフィルター条件を設定して、バリアントを選別します。
eupatho-bioinfomatics.hatenablog.com
GATK解説シリーズ パート8 GATK VariantFilterationの使い方
- GATK VariantFiltrationを使って、vcfファイルから低クオリティのバリアントを除外します。
eupatho-bioinfomatics.hatenablog.com
GATK解説シリーズ パート9GATK BaseRecalibratorとApplyBQSRの使い方
- 前回のGATK VariantFilterationで出力された、
merged_snps/indels_filtered.vcfを基にBQSRを行います。
eupatho-bioinfomatics.hatenablog.com
GATK解説シリーズ パート10 GATK BQSR後のVariant Calling
前回のGATK VariantFilterationで出力された、
*.bqsr.bamを基に2回目のVariant Callingを行います。今回行う内容は、GATK解説シリーズ Part 5 からPart 8で行った仕事の繰り返しになります。新しく覚えることは特にありません。
今回得た
merged_snps/indels_filtered.vcfを使って、多様なゲノムワイド解析を行うことができます。
eupatho-bioinfomatics.hatenablog.com
GATK解説シリーズ パート11 GATK FastaAlternateReferanceMakerを使って代替参照配列を作成する
GATK FastaAlternateReferanceMakerを使って、元となった参照配列に変異情報を当てはめる形で代替リファレンス配列(Fasta形式)を取得する。
感覚的にはVCF→FASTAに変換しているが、実際に変換したわけでは無く、VCFファイルに含まれる代替対立遺伝子(alternate allele)の配列をリファレンス配列と交換することで、変異を取り込んだ新しいFASTAを得ている。 ゲノムを新たにアセンブリしたわけではないので、利用には少し注意が必要。
自分で使用して失敗してしまった注意点もお伝えしますので、初めて使う方はぜひ読んでから開始することをお勧めします!
GATK Forumの説明を読んだだけで実行すると、十中八九同じ失敗をすると思います。
eupatho-bioinfomatics.hatenablog.com
SNPを使った解析の実践例 その1
eupatho-bioinfomatics.hatenablog.com
参考サイト
GATKの公式HP
https://gatk.broadinstitute.org/hc/en-us
初めてGATKに触れる時に、非常にお世話になったサイト