MultiQCで分析結果をまとめてみる。[MultiQC]
MultiQCの使い方
導入難易度★☆☆☆☆
使用難易度★☆☆☆☆
このツールで何ができる?
MultiQCは、他のバイオインフォマティクスツールで生成された結果やログファイルを要約するレポートツールです。
MultiQCを起動すると、指定されたファイルパスを検索して適合するファイルを自動で見つけます。MultiQCは、これらのファイルから情報を抜き出し、単一のHTMLレポートファイルを生成します。また、解析されたすべてのデータを含むフォルダを生成します。
私の使用感
品質管理の必須ツールとして利用しています。
一つ一つの結果を細かく見る前に、全体の結果を把握するのに重宝します。
Twitterで記事の更新をお知らせしているので、興味を持たれた方は是非フォローをお願いします。
フォローする @harrykun_blog
1. ツールのインストール
conda install -c bioconda -c conda-forge multiqc
インストールが正しく導入されたか確認する。
multiqc --help
正しく導入されていれば、下記が表示される。
Usage: multiqc [OPTIONS] <analysis directory> MultiQC aggregates results from bioinformatics analyses across many samples into a single report. It searches a given directory for analysis logs and compiles a HTML report. It's a general use tool, perfect for summarising the output from numerous bioinformatics tools. To run, supply with one or more directory to scan for analysis results. To run here, use 'multiqc .' See http://multiqc.info for more details. Author: Phil Ewels (http://phil.ewels.co.uk)
eupatho-bioinfomatics.hatenablog.com
2. 解析可能なツールの一覧
詳細は、公式HPを参照のこと。
Adapter Removal
AfterQC
bcl2fastq
BioBloom Tools
Cluster Flow
Cutadapt
ClipAndMerge
FastQ Screen
FastQC
Fastp
FLASh
Flexbar
InterOp
iVar
Jellyfish
KAT
leeHom
minionqc
pycoQC
Pychopper
SeqyClean
Sickle
Skewer
SortMeRNA
Trimmomatic
BISCUIT
Bismark
Bowtie 1
Bowtie 2
BBMap
HiCUP
HiC-Pro
HISAT2
Kallisto
Longranger
Salmon
STAR
TopHat
DRAGEN
MALT
Bamtools
Bcftools
biobambam2
BUSCO
bustools
Conpair
DamageProfiler
DeDup
deepTools
Disambiguate
featureCounts
Fgbio
GATK
goleft indexcov
Hap.py
HiCExplorer
HOPS
HOMER
HTSeq
JCVI Genome Annotation
Kaiju
Kraken
MACS2
methylQA
mosdepth
miRTrace
mirtop
MTNucRatio
MultiVCFAnalyzer
ngsderive
OptiType
phantompeakqualtools
Peddy
Picard
Preseq
Prokka
PURPLE
qc3C
QoRTs
Qualimap
QUAST
RNA-SeQC
Rockhopper
RSEM
RSeQC
Samblaster
Samtools
Sentieon
Sargasso
Sex.DetErrMine
EigenStratDatabaseTools
Slamdunk
SnpEff
SNPsplit
Somalier
Supernova
Stacks
THeTA2
VarScan2
VCFTools
VEP
VerifyBAMID
なお、本ブログの過去記事↓
eupatho-bioinfomatics.hatenablog.com
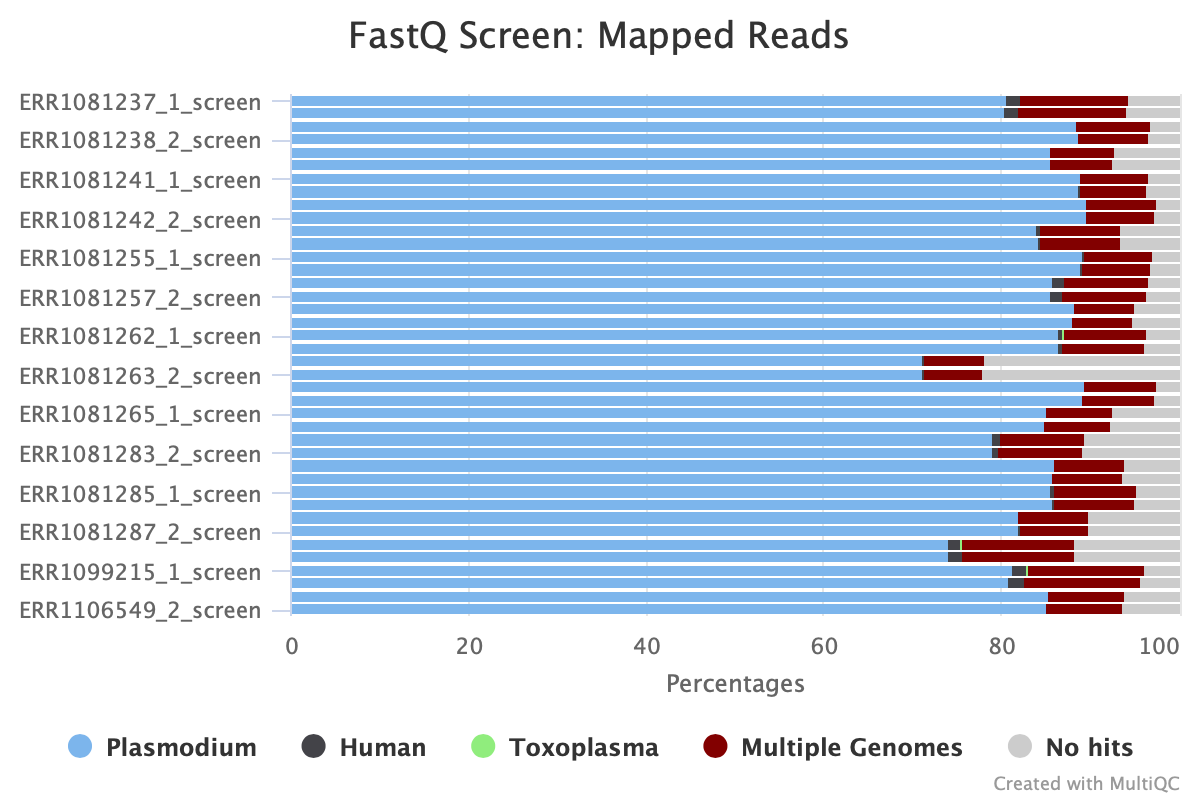
で作成したFastQ screenの結果を使用する。
3. 解析の準備に必要な手順
前回(https://eupatho-bioinfomatics.hatenablog.com)解析したFastQ-screenの結果を入れたフォルダに移動する。
cd workingdir/fastQscreen
4. multiqcを実行する。
multiqc .
改正が終了すると、フォルダ内にmultiqc_dataと、multiqc-report.htmlが生成される。
htmlを開くと結果を見ることができる。
今回はこれで終わりです。 よければ他の記事のも見ていってください。